Forschungsschwerpunkte
- Optische, molekulare und serologische Multiparameteranalysen für die klinisch-mikrobiologische Diagnostik und Epidemiologie
- Schwerpunkt: Detektion und Verständnis von Antibiotikaresistenzen pathogener Bakterien
- Assay-basierte Forschungsaktivitäten mit Hilfe modernster molekularer Technologien, wie digitaler PCR, real-time PCR, Next-Generation-Sequenzierung (NGS), Mikroarray-basierte und isothermale Analyseverfahren
- Mikroarray-basierte Muliparameterserologie mittels Antikörperarrays, Antigenarrays und Peptidarrays
- Bioinformatik für das molekulare Assaydesign und der Auswertung entsprechender Daten (z.B. NGS)
- Translation bestehender und neuer Forschungsergebnisse in real verfügbare Produkte in Kooperation mit entsprechenden Partnern
- Aufbau einer „Brücke“ zwischen akademischer Forschung zur Entwicklung neuer innovativer Assays mittels Systemtechnologie und Produktentwicklung und -realisierung mit kooperierenden Firmen der Diagnostikindustrie
DNS-basierte Multiparameterverfahren
Die Abbildung zeigt ein typisches Mikroarraybild nach erfolgter Prozessierung. Die automatisierte Analyse der dunklen Spots wird im Analyzer (l.) durchgeführt.
Foto: AG EhrichtDie Fachabteilung Ehricht hat ihre Arbeit am IPHT im Januar 2019 aufgenommen und vorher, in den vergangenen Jahren, wesentliche Erkenntnisse bei der Entwicklung und Anwendung verschiedenster molekularer Verfahrung für die Detektion, die Epidemiologie und das Verständnis von pathogenen Bakterien und deren Antibiotikaresistenzen (MRSA, ESBL, CRE, VRE u.a.) gewonnen. Dabei kamen vor allem Mikroarray-basierte Verfahren und eigens dafür entwickelte Plattformen zum Einsatz (Abbildung links), die in ihrer gesamten Breite über bioinformatisches Assaydesign, Oberflächenchemie, Fertigung, Testentwicklung, Bild- und Datenauswertung sowie spezifische Anwendungen reichte. Besonderes Augenmerk wurde einerseits auf die Implementierung dieser Forschungsergebnisse in real existierende Produkte und deren Evolution gelegt. Andererseits war ein Hauptfokus, Strategien und Verfahren für die optimale Probenvorbereitung zu erarbeiten und in komplexe Abläufe zu implementieren.
- Braun SD, Jamil B, Syed MA, Abbasi SA, Weiss D, Slickers P, Monecke S, Engelmann I, Ehricht R: Prevalence of carbapenemase-producing organisms at the Kidney Center of Rawalpindi (Pakistan) and evaluation of an advanced molecular microarray-based carbapenemase assay. Future Microbiol 2018, 13:1225-1246.
- Monecke S, Slickers P, Gawlik D, Muller E, Reissig A, Ruppelt-Lorz A, Akpaka PE, Bandt D, Bes M, Boswihi SS et al: Molecular Typing of ST239-MRSA-III From Diverse Geographic Locations and the Evolution of the SCCmec III Element During Its Intercontinental Spread. Front Microbiol 2018, 9:1436.
- Weiss D, Gawlik D, Hotzel H, Engelmann I, Mueller E, Slickers P, Braun SD, Monecke S, Ehricht R: Fast, economic and simultaneous identification of clinically relevant Gram-negative species with multiplex real-time PCR. Future Microbiol 2019, 14:23-32.
Next-Generation-Sequenzierung – Ein Werkzeug zur Entwicklung von Schnelltests
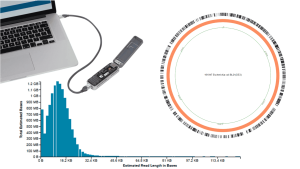
Der von Nanopore entwickelte MinION (o.l.) schafft es durch seine neue porenbasierende Sequenziermethode Genomreads mit Längen bis zu 100kB zu generieren (Diagramm unten). Dadurch ist es möglich Vollgenome schnell und ohne Lücken zu assemblieren (z.B. E. coli BL21).
Foto: AG EhrichtDie von Oxford Nanopore Technologies (Oxford, UK) entwickelte Plattform MinION wird mit ihrer Porentechnologie als der nächste Schritt der Next-Generation-Sequenzierung betrachtet (Abbíldung rechts). Die MinION Technologie basiert auf membrangebundene Protein-Nanoporen. Liegt eine Spannung an dieser Membran an, wird ein Ionenstrom durch die Poren generiert. Passiert ein DNA-Strang die Pore kommt es zu Spannungsänderungen, welche gemessen und später zu Nukleotiden umgerechnet werden. Je Pore können bis zu 400 Nukleotide pro Sekunde gemessen werden, was pro Genom zu Ausgaben von bis zu zwei Gigabasen führen kann. Einzelne Reads können Längen von 50.000-100.000 Basen erreichen. Daher ist es möglich speziell bakterielle Genome einfach und schnell zu analysieren und zu charakterisieren.
Was die Plattform so attraktiv macht, ist ihr vergleichbar günstiger Preis und die kompakte Größe. Die AG Ehricht will daher diese Technologie nutzen, neue bahnbrechende Assays auf molekularer Basis zu entwickeln. Allerdings werden auch andere Methoden der Next-Generation-Sequenzierung (z.B. MySeq, Illumina) etabliert und angewandt.
- Stieber B, Sabat A, Monecke S, Slickers P, Akkerboom V, Muller E, Friedrich AW, Ehricht R: PVL overexpression due to genomic rearrangements and mutations in the S. aureus reference strain ATCC25923. BMC Res Notes 2017, 10(1):576.
Protein- und Peptid-basierte Multiparameterverfahren
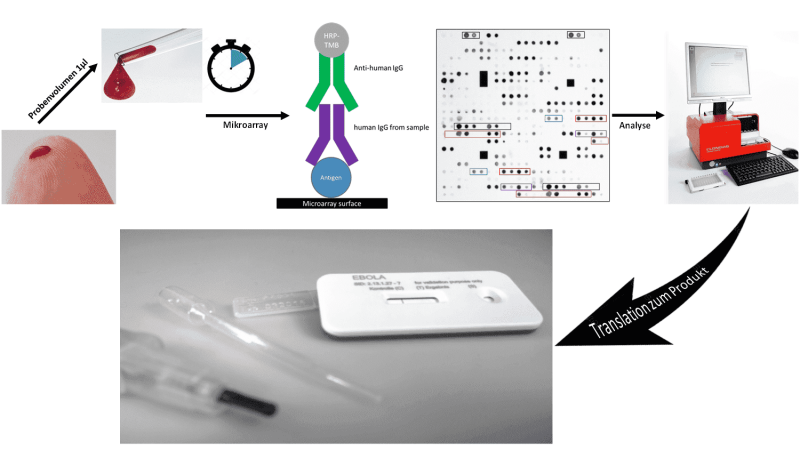
Routinemäßig werden Tests für verschiedene Antigene getrennt und nacheinander durchgeführt, was zu hohen Kosten und zeitlich verzögerten Ergebnissen führt. Außerdem wird eine relativ große Menge an Probe benötigt. Die AG Ehricht konnte belegen, dass die Multiparameter-Serologie mit Protein- und Peptidmikroarrays realisierbar ist. So konnte der IgG-Status und Impfstatus für 30 Erreger parallel und aus 1 µl und einer Gesamtzeit von 2h bestimmt werden (Abbildung unten). Das gleiche Verfahren könnte in Zukunft nicht nur für diagnostische Zwecke, sondern auch für die Testentwicklung (Antigen- und Antikörperscreening) eingesetzt werden. Das Panel der Antigene kann schnell an andere Anwendungen angepasst werden und auch auf Point-of-Care Plattformen wie Lateral Flow übertragen werden.
- Ehricht R, Adelhelm K, Monecke S, Huelseweh B: Application of protein arraytubes to bacteria, toxin, and biological warfare agent detection. Methods Mol Biol 2009, 509:85-105.
-
Sachse K, Rahman KS, Schnee C, Muller E, Peisker M, Schumacher T, Schubert E, Ruettger A, Kaltenboeck B, Ehricht R: A novel synthetic peptide microarray assay detects Chlamydia species-specific antibodies in animal and human sera. Sci Rep 2018, 8(1):4701.
Testprinzip der Multiparameterserologie mit Hilfe von Protein- und Peptidmikroarrays. Ergebnisse der Multiparametertests können schnell in Anwendungen wie dem Lateral Flow eingehen.
Foto: AG Ehricht